
P07 - Thomas Stamminger
Räumlich-zeitliche Kontrolle des transkriptionellen Silencing des menschlichen Cytomegalovirus während der lytischen und latenten Infektion
- Human Cytomegalovirus
- Silencing factors
- Repressor occupancy of viral genome
- Transcriptional signature dictating silencing
- Visualization of viral genome silencing
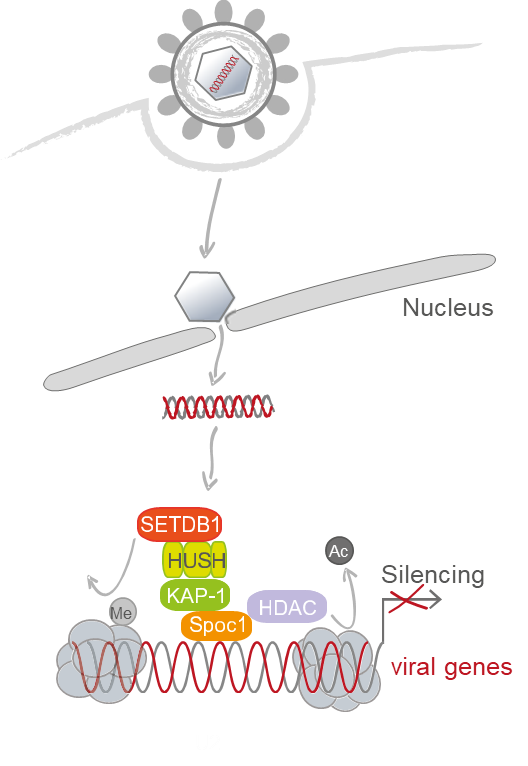
Das humane Cytomegalovirus (HCMV), ein speziesspezifisches Beta-Herpesvirus, persistiert als subklinische, lebenslange Infektion im normalen menschlichen Wirt. Unter Bedingungen einer unreifen oder geschwächten Immunabwehr führt die Reaktivierung aus der Latenz jedoch häufig zu schweren Erkrankungen. Frühere Studien haben gezeigt, dass das HCMV-Genom sowohl während der lytischen als auch der latenten Infektion ein transkriptionelles Silencing erfährt, das durch zelluläre Heterochromatinbildungsfaktoren vermittelt wird. Bislang sind die molekularen Mechanismen, die das Silencing der HCMV-Genexpression steuern, noch unvollständig definiert. Wir haben gezeigt, dass eine Gruppe von zellulären Restriktionsfaktoren (PML, Sp100, DAXX, ATRX, SPOC1), die sich räumlich und/oder zeitlich reguliert mit dem viralen Genom verbinden, zum transkriptionellen Silencing beitragen. Insbesondere haben wir vor kurzem beobachtet, dass der Chromatin-Remodellierungsfaktor SPOC1 mit dem HCMV-Hauptpromotor interagiert, was mit einer Abschaltung der viralen Genexpression korreliert. Da SPOC1 KAP1 rekrutiert und in Verbindung mit dem Human Silencing Hub (HUSH)-Komplex nachgewiesen werden konnte, wird ein Hauptziel dieses Projekts darin bestehen, den Beitrag dieser Faktoren zur Kontrolle des transkriptionellen Silencing von HCMV sowohl während der permissiven Infektion als auch während der Latenz zu untersuchen. Das zweite Ziel ist die genomweite Analyse der viralen Chromatinzugänglichkeit und der Repressorbesetzung mit Hilfe neuartiger Methoden, die eine effiziente epigenomische Profilerstellung bei kleinen Proben ermöglichen. Die erzielten Ergebnisse sollen helfen zu verstehen, wie die Repressorbesetzung den viralen Chromatinzustand diktiert und folglich den Wechsel zwischen Silencing und Aktivierung der viralen Genexpression bestimmt. Ergänzend dazu werden Einzelzell-RNA-Seq-Untersuchungen durchgeführt, um festzustellen, ob eine spezifische Wirts-Transkriptionssignatur mit der HCMV-Silencing-Transkription assoziiert ist, sowie Live-Cell-Imaging-Experimente, um das Silencing einzelner HCMV-Genome zu visualisieren. In Zusammenarbeit mit anderen Projekten des RU wollen wir einen vergleichenden Blick darauf werfen, wie die Repressorbesetzung unterschiedliche virale Chromatin-Zustände diktiert und folglich das Genexpressionsprofil in verschiedenen DNA-Viren bestimmt.
Zur Beantwortung dieser Fragen setzen wir mehrere Instrumente ein, darunter ATAC-Seq, Chromatin-Immunpräzipitations-Sequenzierung (ChIP-Seq, CUT&Tag), RNA-Seq und Einzelzell-RNA-Seq in geeigneten Infektionsmodellen.
.
Dieses breite Spektrum an Techniken ist nur dank der engen Zusammenarbeit innerhalb von DEEP-DV möglich.


