
P03 - Markus Landthaler
Die Rolle von RNA-bindenden Proteinen bei der Regulierung der Transkription in HSV-1-infizierten Zellen und Organoiden
- Regulation of gene transcription
- Herpes Simplex Virus 1
- Protein-RNA interactions
- Role of RNA binding proteins in virus infection
- RNA binding proteins encoded by HSV-1
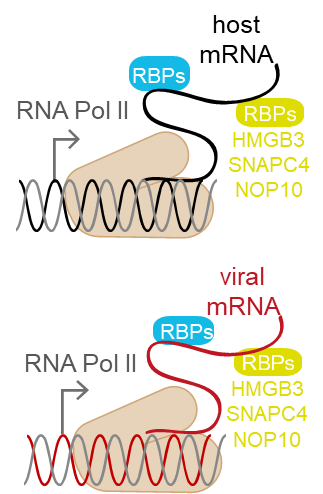
Viren, die sich im Zellkern replizieren, wie z. B. Herpesviren, Polyomaviren oder Adenoviren, beeinflussen den zellulären RNA-Stoffwechsel auf zwei Arten. Erstens muss die virale mRNA aus dem viralen Genom transkribiert werden. Zweitens führt die zelluläre antivirale Reaktion zu transkriptionellen und posttranskriptionellen Veränderungen der Wirtsgenexpression. Im letzten Jahrzehnt wurde deutlich, dass RNA-Moleküle nicht nur ein passives Produkt der Transkription sind, sondern diese selbst auf vielfältige Weise regulieren, beispielsweise über Enhancer-assoziierte RNAs oder Transkriptionsfaktoren-assoziierte nicht-kodierende RNAs. All diese Effekte werden durch RNA-bindende Proteine (RBPs) vermittelt, die somit eine Schlüsselrolle bei der Regulierung der Transkription spielen. Zelluläre und virale RBPs bilden somit eine Schlüsselebene der Genexpression bei Virusinfektionen. Wichtig ist, dass viele interferonstimulierte Gene (ISGs) für RBPs kodieren, wie z. B. die IFIT-Familie, mehrere RNA-Helikasen, Mitglieder der OAS-Proteinfamilie oder splicing-assoziierte Proteine. Daher nehmen wir an, dass RBPs wichtige Modulatoren der viralen Transkription sowie der Transkription des antiviralen Programms des Wirts sind. In der ersten Förderperiode werden wir RBPs in der Wirtszelle identifizieren, die bei der HSV-1-Infektion eine Rolle spielen, sowie RBPs, die im viralen Genom kodiert sind, indem wir unvoreingenommene proteomische Methoden anwenden. Die Bedeutung der identifizierten Wirtszell-RBPs für das Fortschreiten der Infektion wird in HEK293-Zellen und menschlichen Hirnorganoiden mit Funktionsverlust-Experimenten untersucht. Für ausgewählte zelluläre und virale RBPs werden Ziel-Transkripte identifiziert und die Auswirkungen von RBPs auf die Transkription und Chromatin-Konformation untersucht.
Zur Beantwortung dieser Fragen setzen wir unterschiedliche experimentelle Ansätze ein, die auf Hochdurchsatz-Sequenzierung und Datenanalyse beruhen, z. B. mRNA-Interactome Capture, PAR-CLIP, SLAM-Seq, 4SU-Seq und ChIP-Seq.
Dieses breite Spektrum an Techniken ist nur dank der engen Zusammenarbeit innerhalb von DEEP-DV möglich.


