
P02 - Lars Dölken / Florian Erhard
Regulierung und Herpes-Simplex-Virus 1 Gegenregulierung der transkriptionellen Bursting-Kinetik in der frühen Typ I Interferonreaktion
- Systems biology
- Interferon response
- Herpes simplex virus
- Single cell analysis
- Transcriptional bursts
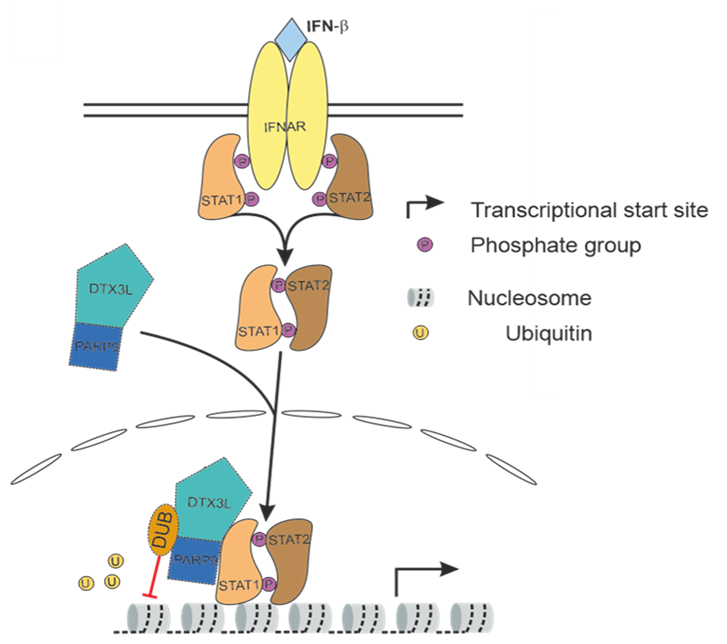
Interferone vom Typ I (IFN) induzieren die Expression von Hunderten von Genen, die die Kontrolle über eindringende Viren verstärken. Eine effiziente Umgehung dieser Gene ist entscheidend für eine produktive Virusinfektion und langfristige Persistenz. Wir entdeckten, dass die Induktion von interferonstimulierten Genen (ISGs) in den ersten zwei Stunden einer Zytomegalovirus-Infektion vor allem auf eine erhöhte Wahrscheinlichkeit von Transkriptionsschüben (Bursts) zurückzuführen ist. Das Ausmaß der Bursts blieb jedoch im Vergleich zur Basalexpression der ISGs in nicht infizierten Zellen unverändert. Kürzlich wurde gezeigt, dass der DTX3L/PARP9-Ubiquitin-Ligase-Komplex an den STAT1/STAT2/IRF9 (ISGF3)-Komplex bindet und Histone innerhalb der ISG-Promotoren ubiquitiniert. Hierdurch wird die Expression einer großen Untergruppe von ISGs verstärkt. Interessanterweise fanden wir heraus, dass das große HSV 1-Tegumentprotein pUL36 mit seiner N-terminalen deubiquitinierenden (DUB) Domäne DTX3L/PARP9 angreift und zwei DTX3L/PARP9 zugeschriebene Ubiquitin-vermittelte Funktionen hemmt, nämlich die Induktion von ISGs und die Rekrutierung von p53-Bindungsprotein 1 (53BP1) an DNA-Schäden. Wir verfolgend jetzt die Hypothesen auf, dass (i) DTX3L/PARP9, welches über den ISGF3-Komplex an ISG-Promotoren rekrutiert wird, zur Umschaltung von ISG-Promotoren von einem nicht-permissiven in einen permissiven Zustand führt und dadurch häufigere Transkriptionsschübe induziert, und (ii) dass das HSV-1 pUL36 DUB dem entgegenwirkt, um die produktive Infektion zu verstärken. Das Hauptziel dieses Projekts ist es, die mechanistische Rolle des pUL36 DUB auf die DXT3L/PARP9-ISGF3-Interaktion bei der Steuerung der genspezifischen Transkription von ISGs aufzuklären. Die gewonnenen Daten werden einen spannenden neuen zellulären Mechanismus beleuchten, durch den ISGs auf der Ebene des transkriptionellen Bursting reguliert werden und wie HSV-1 diesen Mechanismus stört. Schließlich werden wir unseren scSLAM-seq-Ansatz anwenden und neue bioinformatische Methoden entwickeln, um die Kinetik des ISG-Burstings auf Einzelzellebene zu untersuchen. Zusammenfassend werden wir die virale Manipulation der ISG-Induktion untersuchen, um neue zelluläre Mechanismen aufzuklären, die die ISG-Bursting-Kinetik auf Chromatin-Ebene steuern.
Zur Beantwortung dieser Fragen setzen wir ein breites Spektrum modernster Methoden ein, darunter Chromatin-Immunpräzipitationssequenzierung (ChIP-Seq), RNA-seq, Genommanipulation und hochentwickelte Bildgebungsanalysen in geeigneten Infektionsmodellen.
Dieses breite Spektrum an Techniken ist nur dank der engen Zusammenarbeit innerhalb von DEEP-DV möglich.




