Die DEEP-DV Forschungsgruppe (FOR)
Diese von der DFG-geförderte Forschungsgruppe (FOR), bestehend aus MolekularbiologInnen, VirologInnen und BioinformatikerInnen, untersucht die Prozesse der frühen Infektion von Zellen durch DNA-Viren über mehrere Virusfamilien hinweg (Herpesviridae, Adenoviridae, Polyomaviridae) mit modernsten Technologien.

Die einzelnen Projekte sind in zwei Hauptforschungsbereiche (Forschungsbereich 1 und 2) zusammengefasst und werden durch das Projekt Z01 gebündelt, das sich der integrativen funktionellen Genomik der in DEEP-DV untersuchten Infektionskrankheitsmodelle widmet.
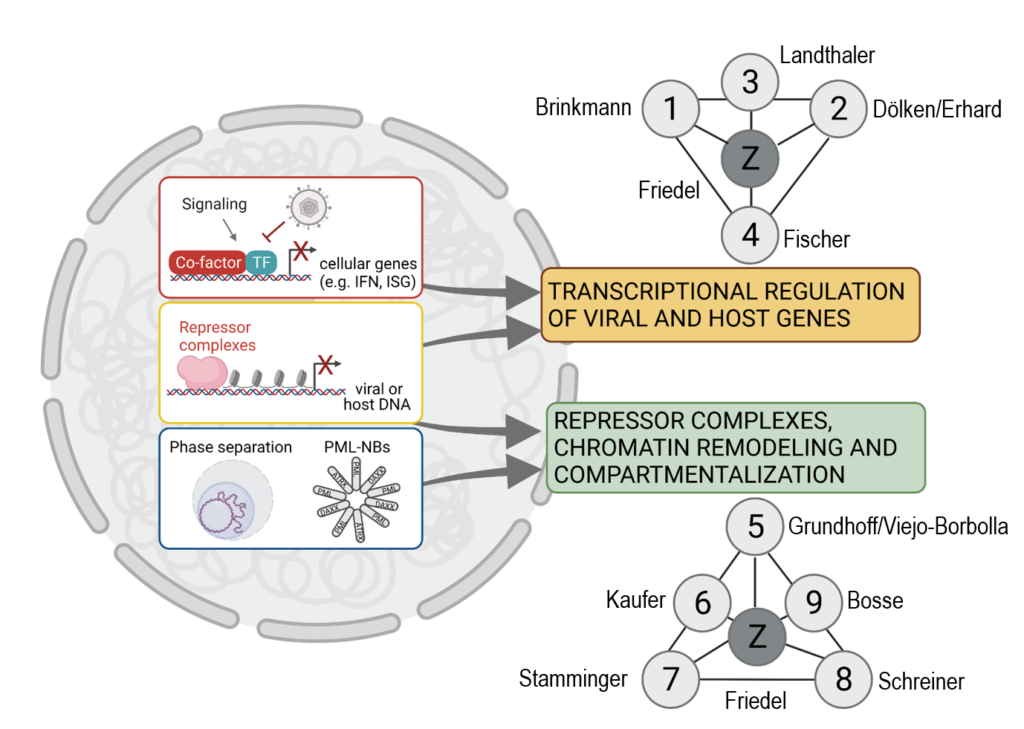
Forschungsbereich 1 (RA1) konzentriert sich auf die transkriptionelle Regulation der Virus- und Wirtsgenexpression zu sehr frühen Zeitpunkten nach dem Eintritt der Viruspartikel in die Wirtszelle. Dazu zählt die durch Mustererkennungsrezeptoren- und Typ I Interferonrezeptoren vermittelte Immunantwort, die zur Expression von Typ I Interferonen (IFN) und IFN-stimulierten Genen (ISG) führt (P01 Brinkmann, P02 Dölken/Erhard, P04 Fischer). RA1 identifiziert außerdem RNA-bindende Proteine viralen und zellulären Ursprungs und untersucht, wie diese Proteine frühe Transkriptionsereignisse nach der Infektion beeinflussen (P03 Landthaler).
Forschungsbereich 2 (RA2) konzentriert sich auf Repressorkomplexe, Chromatin Vorgänge und Kernkompartimente. Die Projekte in RA2 untersuchen synergistisch, wie Viren zelluläre Chromatin-Regulationswege nach dem Eintritt eines naiven viralen DNA-Moleküls in den Kern ausnutzen oder umgehen. P05 Grundhoff/Viejo-Borbolla, P06 Kaufer und P09 Bosse untersuchen, wie der zelluläre Kontext und spezifische Kernkompartimente (1) die lytische Replikation fördern oder (2) zu globalen Chromatin Veränderungen führen können, die entweder das Fortbestehen eines temporär reprimierten viralen Genoms ermöglichen oder zur einer potentiell dauerhaften Inaktivierung viraler Genome führen. Neben der Rolle von globalen Chromatin Veränderungen untersuchen die Projekte P07 Stamminger und P08 Schreiner die Rolle von Repressorkomplexen, die an spezifische genetische Elemente im viralen Genom binden.
Die Expertise der in RA1 und RA2 eingebundenen DEEP-DV-Mitglieder wird durch die bioinformatische Expertise der Friedel-Gruppe (Z01) komplementiert, deren Schwerpunkt auf der funktionellen Genomik von Infektionskrankheitsmodellen liegt. Die Friedel-Gruppe verfügt über umfassende Expertise in der integrativen Analyse funktioneller Transkriptom -und Genomdaten und in der Entwicklung neuer Auswertemethoden mit dem Schwerpunkt auf Virusinfektionen. ContextMap2 ermöglicht beispielsweise das parallele Mapping von Transkriptionsdatensätzen gegen mehrere Genome, wie Wirts- und Virusgenome, und die Identifizierung von Poly-Adenylierungsstellen in Transkripten. Das Workflow-Management-System Watchdog unterstützt die automatisierte Analyse großer funktioneller Genomdatensätze. Mit diesen und in DEEP-DV neu entstehenden Auswertungsmethoden wird Z01 die Daten der einzelnen DEEP-DV Projekte in einem integrativen Vergleich der transkriptionellen und epigenetischen Regulation bei der Infektion mit DNA-Viren zusammenführen. Die Gruppe von Florian Erhard, P02, steuert zusätzliche Expertise in der Einzelzellanalysen und in der Entwicklung von bioinformatischen Methoden bei, die speziell auf Virusinfektionen anwendbar sind.
