Z01 - Caroline Friedel / Florian Erhard
Computergestützte Analyse der Genregulation bei DNA-Virusinfektion
- Integrative multi-omics analyses
- Transcriptional and epigenetic regulation in DNA virus infections
- Promoter-proximal pausing
- Premature and disrupted transcription termination
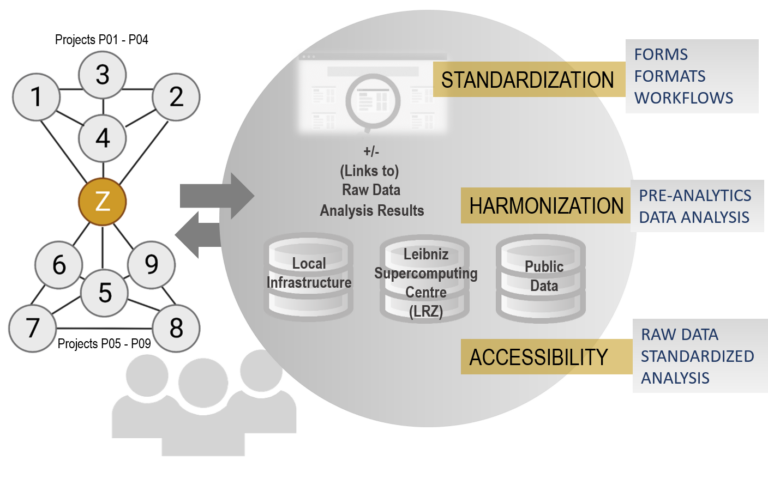
- Standardization of data analysis

In the second funding period, Z01 will develop and analyse the DEEP-DV Transcription Atlas of gene regulation in DNA virus infections at single-cell level. In collaboration with DEEP-DV partners, we will generate a comprehensive, temporally resolved, and harmonized atlas of infection by all DNA viruses studied in DEEP-DV. Due to use of metabolic RNA labelling with 4-thiouridine (4sU), the DEEP-DV Atlas will not only provide snapshots of gene expression throughout infection, but it will also allow dissecting and comparing regulatory processes with unprecedented details. The atlas will be disseminated via a web platform software that enables visualization and analysis of single-cell data from multiple projects and facilitate cross-virus comparisons.
We will also develop new computational approaches to make use of the atlas: We will implement a method to determine the stage of infection of single cells based on viral gene expression patterns. We will extend our previously developed method Heterogeneity-seq for the identification of common and distinct factors that govern infection outcomes for different viruses. Based on this, we will infer gene regulatory networks in DNA virus infections by directly making use of 4sU labelling to differentiate direct regulatory effects from mediating or confounding factors. A particular focus will be on regulation of interferon stimulated genes (ISGs) for which we will compile an up-to-date catalogue of ISGs and their regulation from public RNA- and ChIP-seq data. We will furthermore investigate and cross-compare alternative polyadenylation and aberrant transcription termination in DNA virus infections using the DEEP-DV Atlas. Finally, we will continue our investigations into HSV-1-induced changes in RNA polymerase II pausing and regulation of transposable elements in DNA virus infections together with DEEP-DV partners.
- Integrative multi-omics analyses
- Transcriptional and epigenetic regulation in DNA virus infections
- Promoter-proximal pausing
- Premature and disrupted transcription termination
- Standardization of data analysis

In der zweiten Förderperiode wird Z01 den DEEP-DV-Transkriptionsatlas zur Genregulation bei DNA-Virusinfektionen auf Einzelzellniveau entwickeln und analysieren. In Zusammenarbeit mit den DEEP-DV-Partnern werden wir einen umfassenden, zeitlich aufgelösten und harmonisierten Atlas der Infektion durch alle in DEEP-DV untersuchten DNA-Viren erstellen. Durch die Verwendung der metabolischen RNA-Markierung mit 4-Thiouridin (4sU) wird der DEEP-DV-Atlas nicht nur Momentaufnahmen der Genexpression während der gesamten Infektion liefern, sondern auch die Analyse und den Vergleich regulatorischer Prozesse in bisher nicht dagewesenem Detail ermöglichen. Der Atlas wird über eine Webplattform verbreitet, die die Visualisierung und Analyse von Einzelzelldaten aus mehreren Projekten ermöglicht und virusübergreifende Vergleiche erleichtert.
Wir werden auch neue rechnergestützte Ansätze entwickeln, um den Atlas zu nutzen: Wir werden eine Methode entwickeln, mit der sich das Infektionsstadium einzelner Zellen anhand der viralen Genexpressionsmuster bestimmen lässt. Wir werden unsere zuvor entwickelte Methode Heterogeneity-seq erweitern, um Faktoren zu identifizieren, die den Ausgang der Infektion für verschiedene Viren entweder in gleicher oder unterschiedlicher Weise beeinflussen. Darauf aufbauend werden wir genregulatorische Netzwerke für DNA Virusinfektionen inferieren, indem wir direkt die 4sU-Markierung nutzen, um direkte regulatorische Effekte von indirekten Faktoren zu unterscheiden. Ein besonderer Schwerpunkt liegt auf der Regulierung interferonstimulierter Gene (ISGs), für die wir einen aktualisierten Katalog von ISGs und ihrer Regulierung aus öffentlichen RNA- und ChIP-seq-Daten zusammenstellen werden. Darüber hinaus werden wir alternative Polyadenylierung und aberrante Transkriptionsterminierung in DNA Virusinfektionen mit Hilfe des DEEP-DV Atlas vergleichend untersuchen. Schließlich werden wir gemeinsam mit den DEEP-DV-Partnern unsere Untersuchungen zu HSV-1-induzierten Veränderungen in der RNA-Polymerase II Pausierung und der Regulierung von Transposons in DNA Virusinfektionen fortsetzen.